...
| English |
|---|
An XBRL taxonomy technically consists of different XSD and XML files that are linked to each other. A so-called "entry point" represents the unique entry point to an XBRL taxonomy. The XBRL Portal allows the administration of different entry points as well as the storage of XSD and XML files in a cache. The taxonomies are published on the Internet and are usually a ZIP compressed collection of XSD and XML files. For example from EBA and EIOPA:
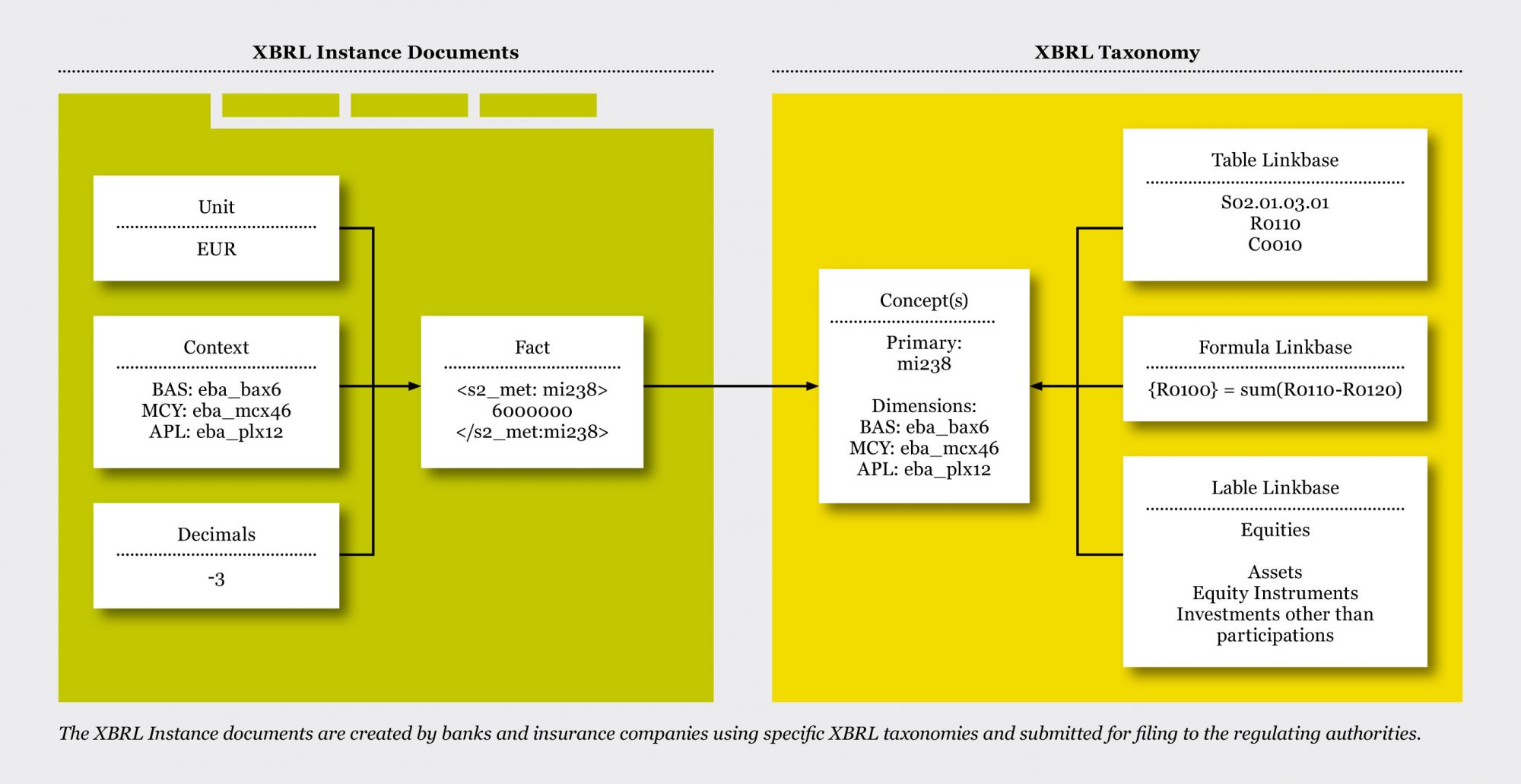
The so-called entry point is one of the files that loads all others. It defines for the reporting entities, which taxonomies are to be used for which report on which due date. For Example (PDF Accepted Entry Points): A filing, which is an XBRL file, is based on an XBRL taxonomy. The taxonomy defines the elements of which the filing can consist as well as their meaning and relationships to each other. In the XBRL filing itself, however, only values that refer to an element from the taxonomy are defined. Furthermore, the taxonomy contains validation rules and information for displaying the data in tabular form. An XBRL filing must be valid for the corresponding XBRL taxonomy, otherwise it can also be marked as "rejected". The definition of the validity of a taxonomy also affects the use of this taxonomy. When a filing is received, it is only accepted if the due date used is within the validity limits of the taxonomy. On the XBRL Portal taxonomies are not a static component of the software, but can be dynamically loaded and processed. A new taxonomy can easily be imported and used immediately. No changes to the software are necessary. On the left side you see an XBRL filing, on the right an XBRL taxonomy. The XBRL taxonomies are among the most important configurations of the XBRL portal, along with the reporting requirements. They must be set up correctly before XBRL filings can be created, submitted or analysed. |
...
| German |
|---|
Eine XBRL-Taxonomie besteht technisch gesehen aus verschiedenen XSD- und XML-Dateien, welche untereinander verlinkt sind. Ein sogenannter „Entry Point" stellt den eindeutigen Einstiegspunkt zu einer XBRL-Taxonomie dar. Das XBRL Portal erlaubt sowohl die Verwaltung von verschiedenen Einstiegspunkten als auch die Hinterlegung der XSD- und XML-Dateien in einem Cache.
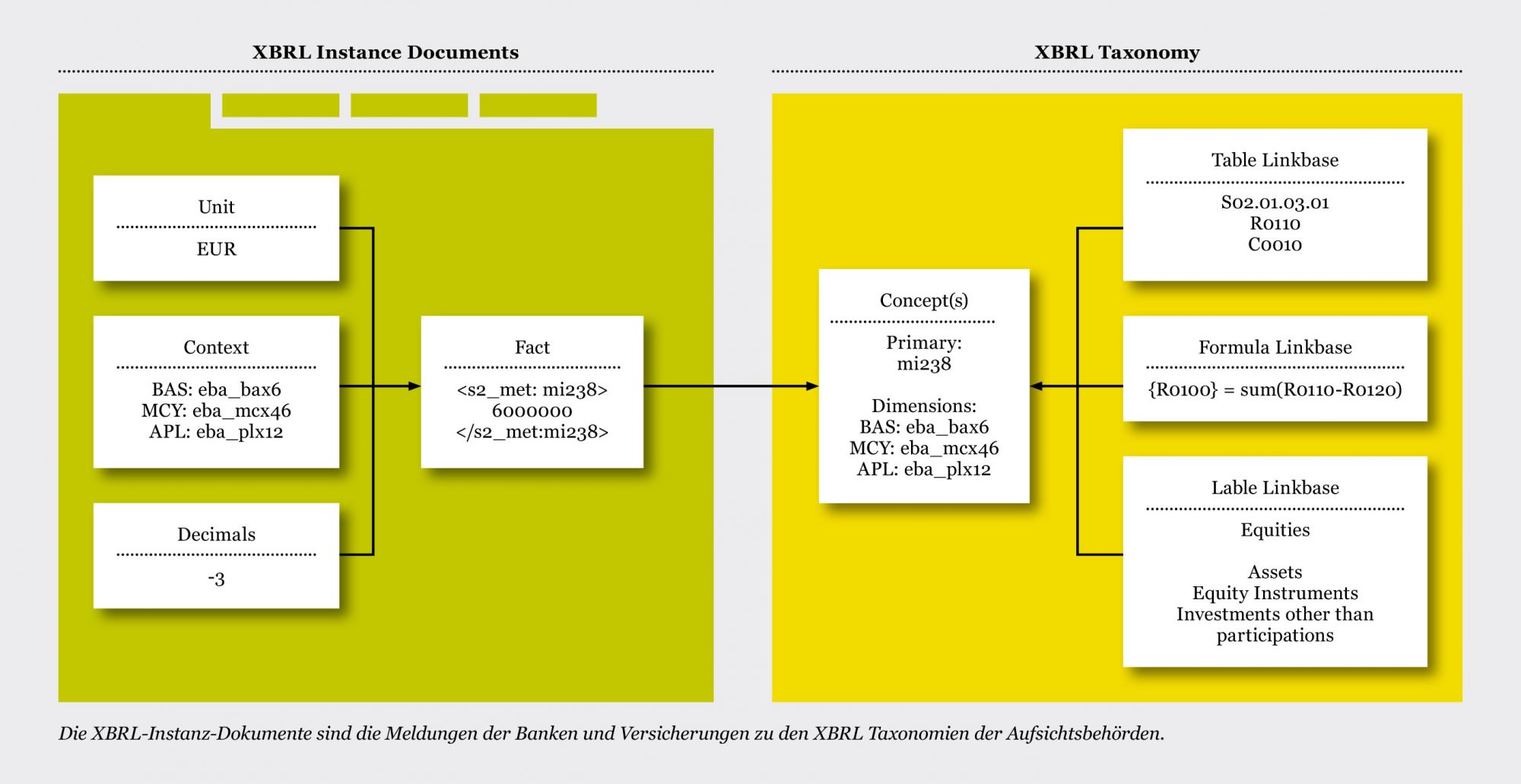
Der sogenannten Entry Point (Einstiegspunkt) stellt eine der Dateien dar, welche alle anderen nachlädt. Es wird für die meldenden Entities definiert, welche Taxonomien zu welchem Stichtag für welche Meldung verwendet werden sollen. Beispielsweise (PDF Akzeptierte Entry Points): Eine Meldung, also eine XBRL-Datei, basiert auf einer XBRL-Taxonomie. Die Taxonomie definiert dabei die Elemente, aus denen die Meldung bestehen kann sowie deren Bedeutung und Beziehungen untereinander. In der XBRL-Meldung selber werden dagegen nur Werte definiert, die auf ein Element aus der Taxonomie verweisen. Weiterhin enthält die Taxonomie Validierungsregeln und Informationen zur Darstellung der Daten in Tabellenform. Eine XBRL-Meldung muss valide gegen die entsprechende XBRL-Taxonomie sein, ansonsten kann diese auch als „abgelehnt" dargestellt werden. |
...